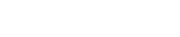文献解读 | 机器学习结合代谢组学,挖掘胃癌诊断及预后标记物
胃癌(Gastric Cancer,GC)是全球癌症相关死亡率的主要原因之一,迫切需要开发早期检测策略和精确的术后干预措施。然而,目前用于早期诊断和患者风险分层的无创生物标志物的识别仍然不足。
本研究的目标是开发一种基于代谢组学的机器学习预测模型,用于胃癌的早期诊断和预后评估,以期提高临床治疗的及时性和精确性。
01研究方法
研究队列由389名胃癌患者和313名非胃癌对照构成,共702份血浆样本,使用靶向代谢组学方法,检测了包括氨基酸、有机酸、核苷酸、核苷、维生素、酰基肉碱、胺类和碳水化合物在内的147种代谢物。
质控样本监测仪器稳定性,数据标准化以消除批次间变异,鉴定组间显著差异的代谢物,并进行KEGG通路富集分析。
使用LASSO回归算法进行特征选择,筛选出关键代谢物;利用随机森林模型训练所选代谢物特征,构建胃癌诊断模型,并通过自助法聚合多个决策树以提高准确性;将模型应用于测试集,并使用接收者操作特征曲线下面积和敏感性/特异性等指标评估模型的诊断性能;构建一个包含多种代谢物的预后模型,并使用随机生存森林方法训练优化。
将临床特征如TNM分期、肿瘤宏观表现和血管肿瘤栓塞等因素纳入预后模型,以评估这些因素对模型预测能力的影响。
02研究结果
开发了一个包含10种关键代谢物的胃癌诊断模型(10-DM模型),在测试集中的敏感性达到了0.854,特异性为0.926,AUROC值为0.967,显示出比传统癌症蛋白标志物更高的敏感性;
模型在诊断早期胃癌(IA/IB期)方面表现出色,对于IA期患者,预测准确率达到90.9%,对于IB期患者达到92.7%,与传统的临床肿瘤标志物(CA19-9、CA72-4和CEA)相比,显示出更高敏感性(0.925 vs.0.428)。
开发了一个包含28种代谢物的预后模型(28-PM模型),该模型在测试集上的AUROC值为0.832,C-index值为0.83,显示出良好的预测能力;预后模型的独立性验证显示,模型中的代谢物与患者的总体生存情况显著相关。
另外,通过单变量Cox回归分析,研究人员比较了临床参数与预后模型,显示一些与预后显著相关的临床变量,包括TNM分期、肿瘤宏观表现和血管肿瘤栓塞,其预测效果均不如28-PM模型。
03研究结论
研究结果表明,通过机器学习结合靶向代谢组学数据,可以有效地识别胃癌的早期诊断和预后预测的生物标志物,为胃癌的精准医疗提供了新的视角和工具。
04研究结果展开
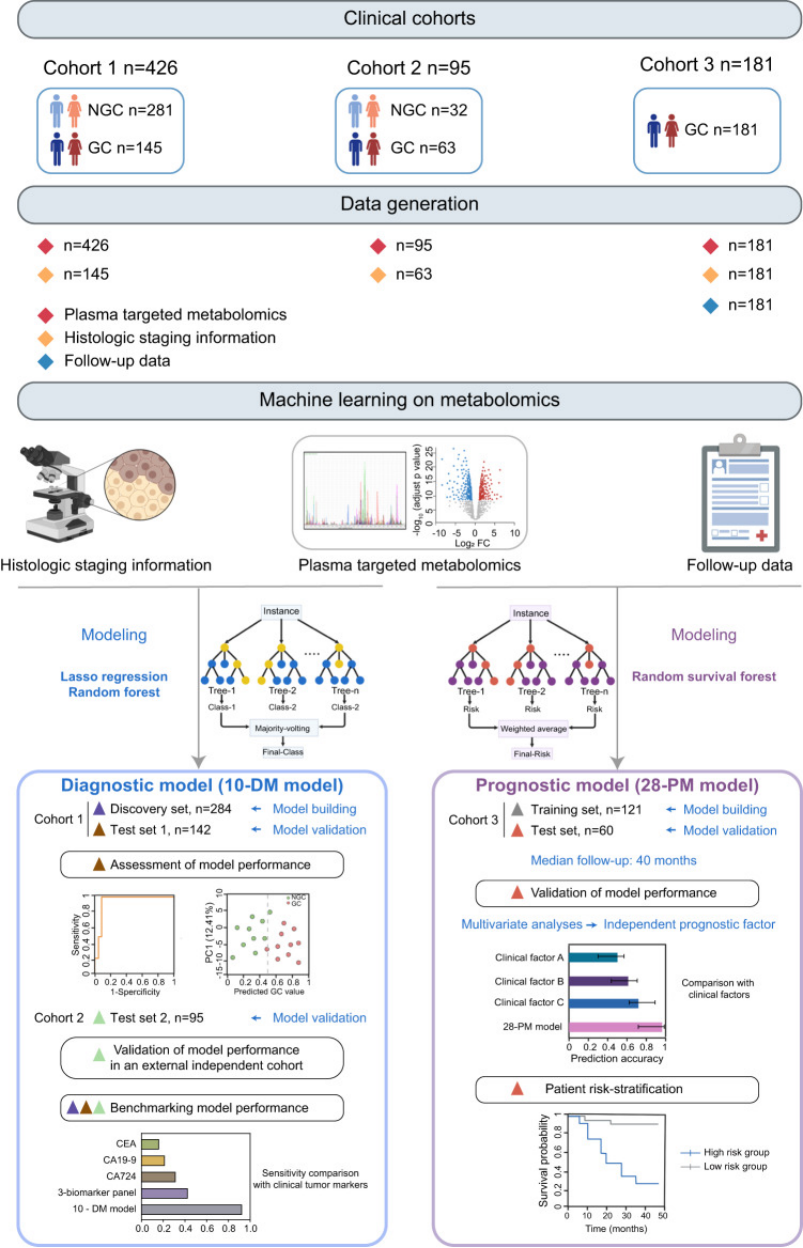
图1.展示了本课题研究整体路线图
包括了数据收集、机器学习建模、模型构建、模型验证以及性能评估步骤。研究包括了三个临床队列(Cohort 1,Cohort 2,Cohort 3),队列1和2包含血浆靶向代谢组学和组织病理学数据,队列3额外包含了随访数据。
利用来自队列1数据进行机器学习创建10-DM模型,模型进一步在队列2中进行验证;利用队列3数据,开发预后模型(28-PM模型)。并将这两个模型与传统临床使用的生物标志物/临床特征进行性能对比。
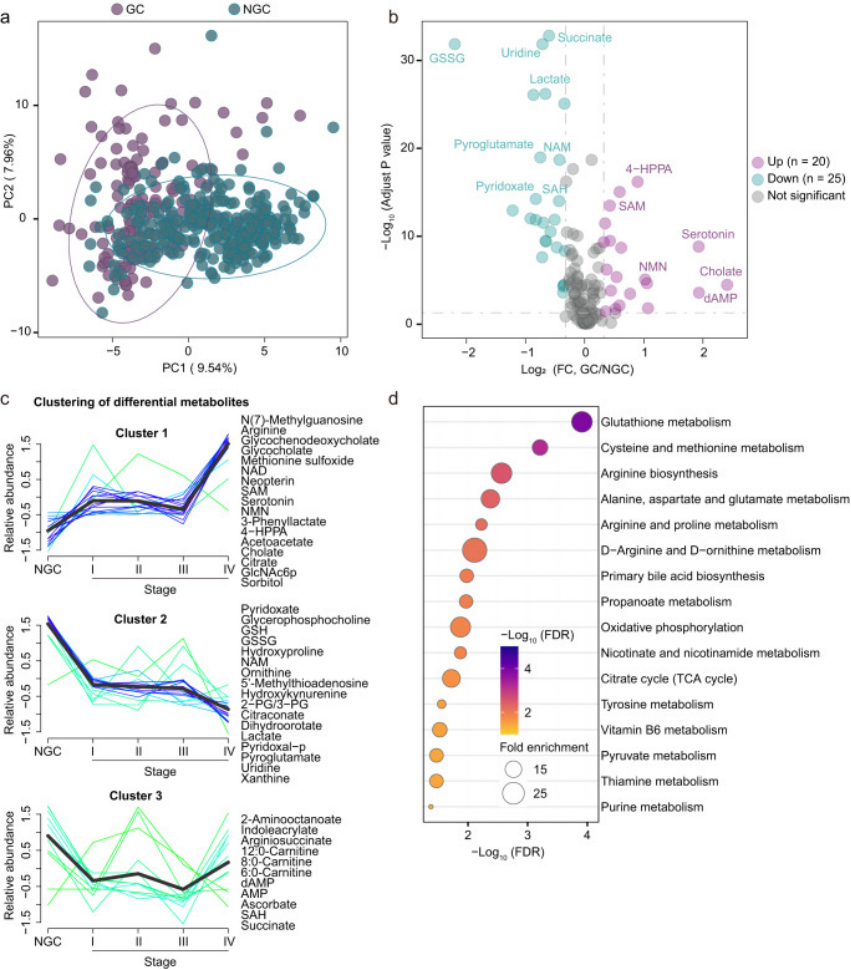
图2展示了胃癌组与对照组在血浆代谢物水平上的差异
a.队列1(n=426)血浆靶向代谢组学数据的主成分分析(PCA),胃癌(紫色)。
b.队列1组间血浆代谢组学数据火山图比较分析,紫色(上调)和绿色(下调)表示。
c.代谢物聚类分析,使用Mfuzz算法对GC进展过程中的代谢物变化轨迹进行了聚类分析,展示了每个聚类中代表性代谢物的变化趋势,聚类中的代表性代谢物标识在右侧。
d.KEGG通路富集分析,展示GC患者和对照之间差异代谢物所在代谢通路。
图3.展示了利用机器学习算法与数据,开发胃癌诊断预测模型的关键步骤和性能评估结果
a.模型设计流程:展示了如何使用最小绝对收缩和选择算子(LASSO)回归算法以及随机森林算法进行特征选择和模型训练,以及10-DM模型在测试集和外部测试集中的验证。
b.ROC曲线分析:展示了在测试集1中,使用接收者操作特征(ROC)曲线评估10-DM模型对胃癌患者诊断的性能。ROC曲线下面积(AUROC)和95%置信区间(CI)提供了模型预测准确性的量化。
c.十个代谢物对模型的贡献:展示了通过LASSO回归算法选出的十个代谢物对胃癌诊断模型的贡献度,包括琥珀酸、尿苷、乳酸、SAM、丙酮酸、2-氨基辛酸、新蝶呤、N-乙酰-D-葡萄糖胺6-磷酸(GlcNAc6P)、5-羟色胺和烟酰胺单核苷酸(NMN)。
d.模型性能评估:通过比较不同数据集中的预测值和实际疾病状态,展示了10-DM模型在区分胃癌患者和非胃癌对照组(NGC)方面的性能。
e.早期胃癌的诊断能力:特别展示了10-DM模型在诊断早期胃癌(IA/IB期)方面的能力,包括在测试集1和外部测试集2中的预测准确性和敏感性/特异性。
f.与传统方法的比较:展示了10-DM模型与传统的临床肿瘤标志物(如CA19-9、CA72-4、CEA)相比,在诊断胃癌方面的性能提升。
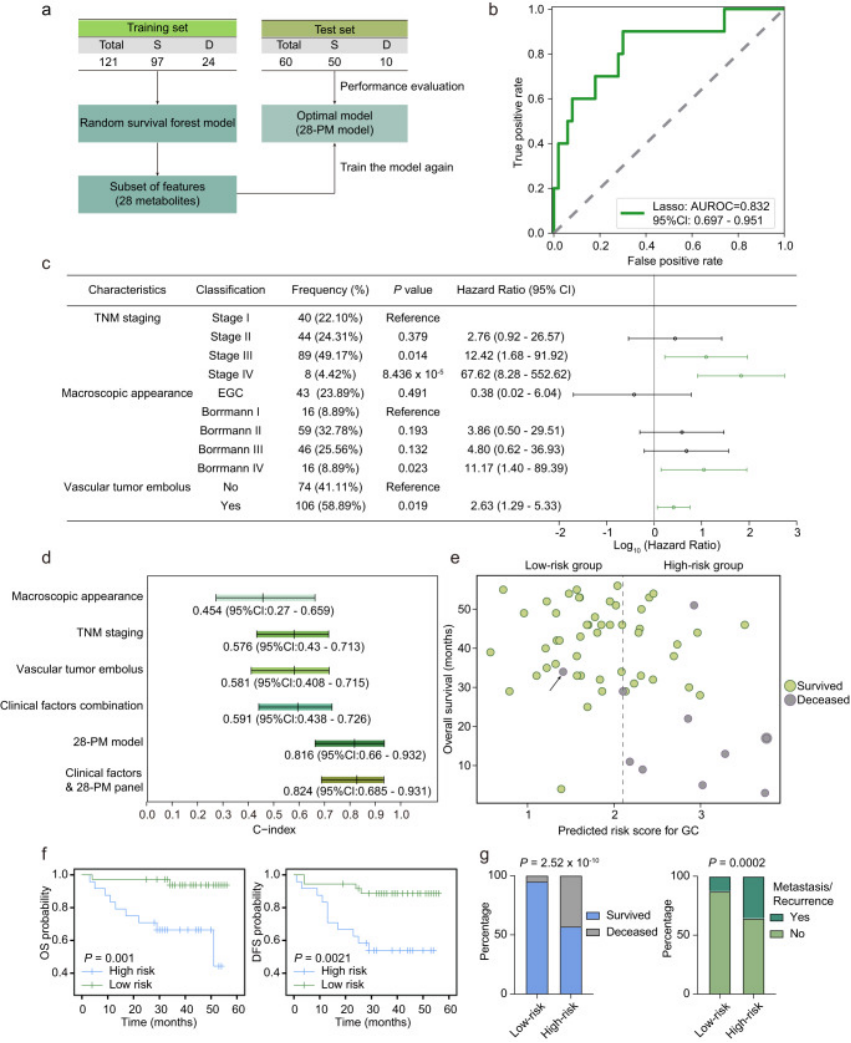
图4展示了预后模型在预测胃癌患者生存结果方面的有效性
a.展示了使用随机生存森林方法建立的28-PM预后模型的设计流程。该流程包括了模型训练、特征选择和性能评估的步骤。
b.通过ROC曲线展示了28-PM模型在测试数据集上对胃癌患者生存结果的预测准确性。曲线下面积(AUROC)和95%置信区间(CI)提供了模型预测性能的量化指标。
c.森林图(Forest plot)显示了通过单变量Cox回归分析识别的与胃癌患者预后显著相关的临床参数,包括TNM分期、肿瘤的Borrmann宏观分型和血管肿瘤栓塞等,并给出了它们的P值和风险比(Hazard Ratio,HR)。
d.比较了28-PM模型与单独的临床参数(如TNM分期、宏观类型、血管肿瘤栓塞)以及这些参数与模型结合使用时的预测效能,C-index用于评估各模型的预测准确性。
e.通过风险评分将测试集中的患者分为高风险和低风险两组,并通过图中的点阵图展示了每位患者的生存状态。
f.展示了根据28-PM模型风险评分分层的胃癌患者的生存曲线,包括总生存期(OS)和无病生存期(DFS),并通过对数秩检验(Log-rank test)计算了P值。
g.展示了高风险和低风险组在患者生存状态和复发/转移情况的分布,以及相应的P值,这些结果表明28-PM模型能够成功识别出需要更精细化治疗的高风险患者群体。
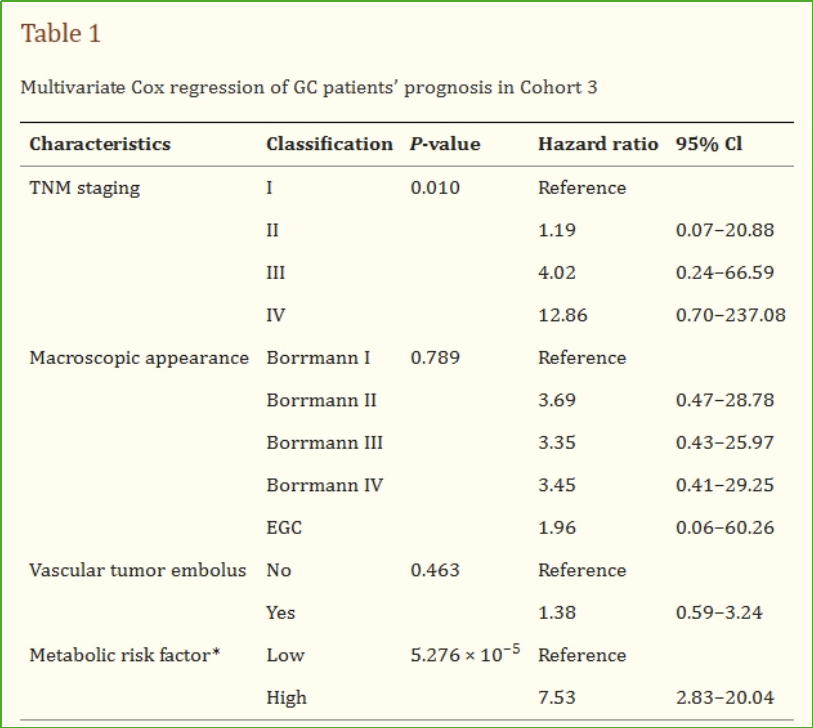
表1.展示多变量Cox回归分析的结果,用于识别胃癌(GC)患者的独立预后因素
相关参数包括:不同临床和代谢特征,包括TNM分期、肿瘤宏观表现、血管肿瘤栓塞,以及由28-PM模型确定的代谢风险因素;其中与预后相关的P值小于0.05为统计显著。
表中给出了特征与患者预后不良的风险比(Hazard Ratio,HR),及95%置信区间(Confidence Interval,CI)。通过多变量分析确定了哪些特征是胃癌患者的独立预后因素,数据表明28-PM模型的代谢风险因素是一个高度显著的独立预后因素。
05总结与启发
该篇文献发表于Nature Communications(IF>16),研究人员使用了多个队列,此策略增强了研究结果的普适性和可靠性,有关检测专注于特定代谢物的靶向分析,提高了疾病相关生物标志物的精确度,使用LASSO回归和随机森林算法等机器学习算法筛选关键代谢物,提升了模型的解释力和实用性,优化了特征选择和模型预测能力。
对于验证环节,采用了系统化模型构建和验证流程,确保了模型的稳定性和临床应用潜力,多变量分析确认了代谢风险因素作为独立预后因素的重要性,结合了相关临床参数,探索了临床参数与机器学习模型的结合,以及对预后评估的增益。
有关性能评估则应用了AUROC、C-index等工具全面评估模型性能,这些分析经验提示了结合机器学习与生物标志物研究的潜能,为临床决策提供支持,并为疾病诊断和预后评估提供了科学依据。
参考文献
Chen Y,Wang B,Zhao Y,et al.(2024).Metabolomic machine learning predictor for diagnosis and prognosis of gastric cancer.Nature Communications,15:1657.https://doi.org/10.1038/s41467-024-46043-y
免疫力|带你了解可以防御“外敌“的免疫基因
免疫力是人体自身的防御机制,可以识别和消灭外来侵入的任何异物(病毒、细菌等),是处理衰老、损伤、死亡、变性的自身细胞和病毒感染细胞的能力。在人体受到外来物刺激之后,免疫系统会被激活,各种免疫相关调控因子介入,使得人体尽快恢复内环境稳定。
2022-05-09 07:53:05
国家药监局科普|牙膏不能治疗疾病
目前,市场上有一些名称冠以“牙膏”的热销产品,宣称具有“抗幽门螺旋杆菌”、“通过抗幽门螺旋杆菌去除口臭”等功效。这些产品很多不是真正意义上的牙膏。那么,什么是牙膏?牙膏能治疗疾病么?这里,提醒广大消费者注意以下几点:
2022-01-12 01:52:43
- 热线电话
- 业务咨询
- 快速询价
- 在线客服
- 报告验证

.jpg)
.jpg)